您好,欢迎光临艾维缔科技怀来有限公司官方网站!
热线:0313-5935520/521
m6A RNA甲基化片段富集试剂盒(qPCR版)【MeRIP试剂盒】
众所周知,N6-甲基腺苷(m6A)是真核生物RNA分子中最常见和最丰富的修饰。由甲基转移酶复合物METTL3催化m6A修饰,并通过M6A RNA去甲基化酶FTO和ALKBH5,以α-酮戊二酸(α-KG)-和Fe2+-依赖的方式催化M6A去甲基化。METTL3、FTO和ALKBH5在许多生物中起着重要作用,从发育和代谢到生育的过程。在所有RNA碱基甲基化中,m6A占80%以上,存在于各种物种中。m6A主要分布在mRNA中同样也发生在非编码RNA中,如tRNA、rRNA和snRNA。在mRNA转录物中m6A的相对丰度已被证明影响RNA代谢过程,如剪接,核输出,翻译能力和稳定性,以及RNA转录。由m6A RNA甲基酶和脱甲基酶缺陷引起的m6A甲基化水平异常可能导致RNA功能障碍并引起疾病。例如,由于FTO突变患者的FTO活性增加,靶m RNA中m6A的异常低水平,通过一种未定义的途径,导致肥胖和相关疾病的发生。对RNA的化学M6A修饰的动态性和可逆性,也可作为一种具有深远生物学意义的新表观遗传标记。因此,更多有用的信息,以更好地理解m6A RNA甲基氧化RNA 转录物的水平和分布有利于疾病的诊断和治疗。目前,有几种方法被用于表位范围的m6A映射。这些方法包括MeRIP,PA-m6A-seq,miCLIP和m6A-CLIP。MeRIP已被广泛应用,但在m6A分析中无法实现高分辨率。PA-m6A-seq、mi CLIP和m6A-CLIP提高了分析分辨率,但由于重复性差和工艺复杂。特别是,这些方法耗时(>2天)和费用昂贵。为了解决这些问题,IVD Technology Corporation联合开发了一种新的方法:CUT&RUN M6A富集【 (利用核酸酶对m6A进行裂解和回收)】。我们的创新方法将 MeRIP 和 m6A-CLIP 的优点与最快的程序结合起来特别的推出了:m6A RNA甲基化片段富集试剂盒(qPCR版)【简称:MeRIP试剂盒】。
 m6A RNA甲基化片段富集试剂盒(qPCR版)【简称:MeRIP试剂盒】 具有以下优势和特点:
m6A RNA甲基化片段富集试剂盒(qPCR版)【简称:MeRIP试剂盒】 具有以下优势和特点:
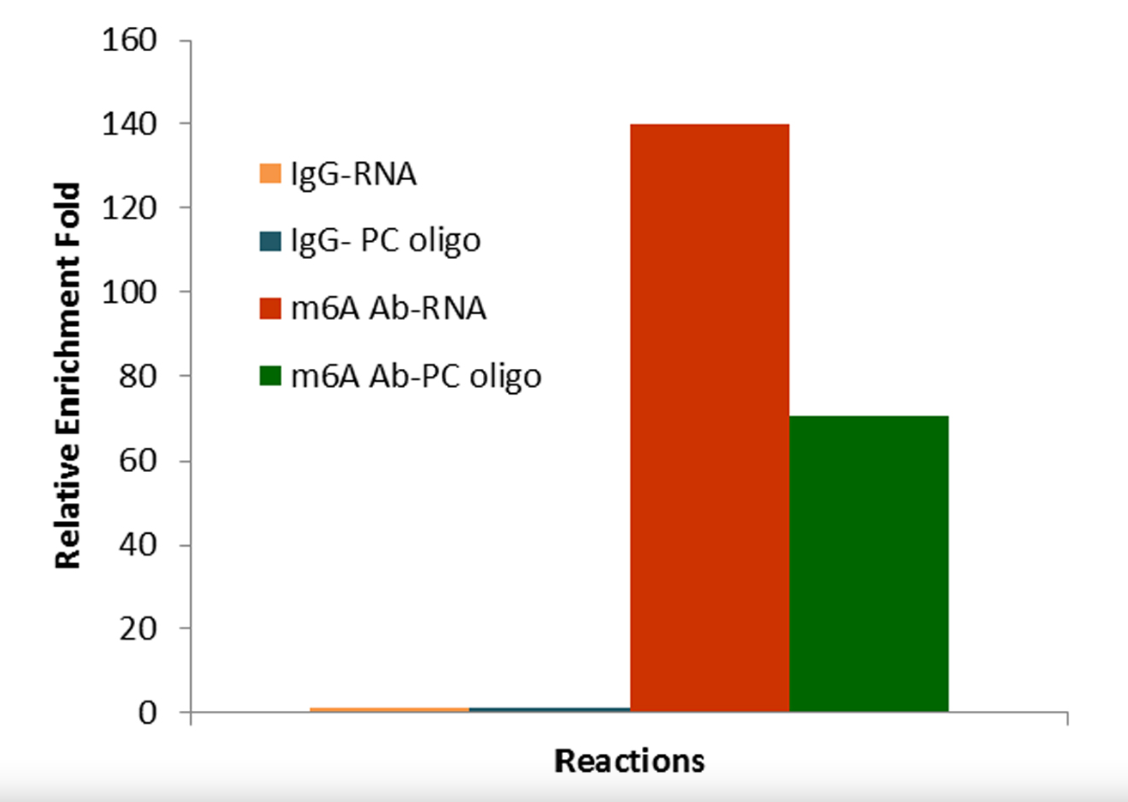
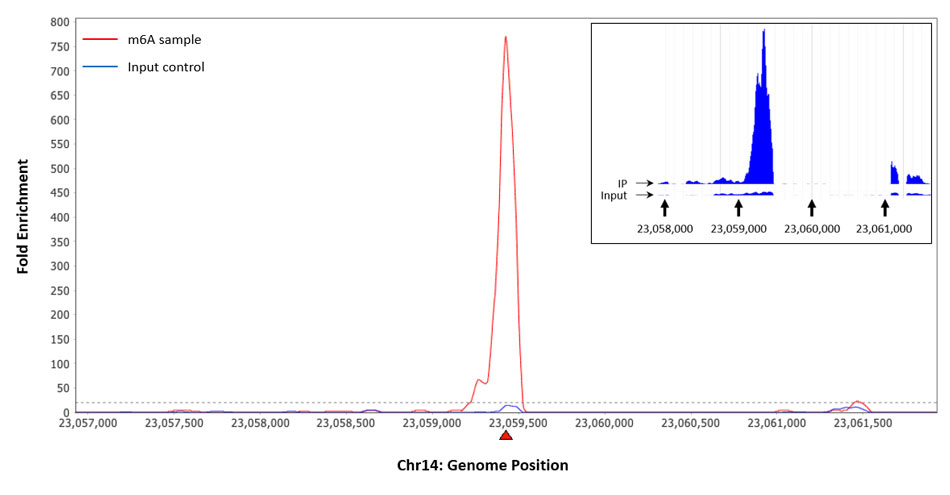
- 高度富集:使用RNA 切割酶混合物同时对含有m6A 的目标序列两端的RNA 进行片段化和切割/移除任何RNA 序列,而不影响被抗体占据的RNA 区域。短片段RNA 仅与抗m6A 抗体结合。因此,非常可靠地确定真正的m6A 富集目标区域,并可实现高分辨率绘图。
- 低输入:不结合的RNA 切割和免疫捕获是在同一个单管中处理的,这样可最大限度地保护含有m6A的目标区域,并最小化样本损失,允许输入RNA 低至500ng。
- 极小背景:在含m6A 目标序列的两(2)端切割非结合RNA 序列,可最小化MeRIP/测序背景,允许1000万读的数据分析。
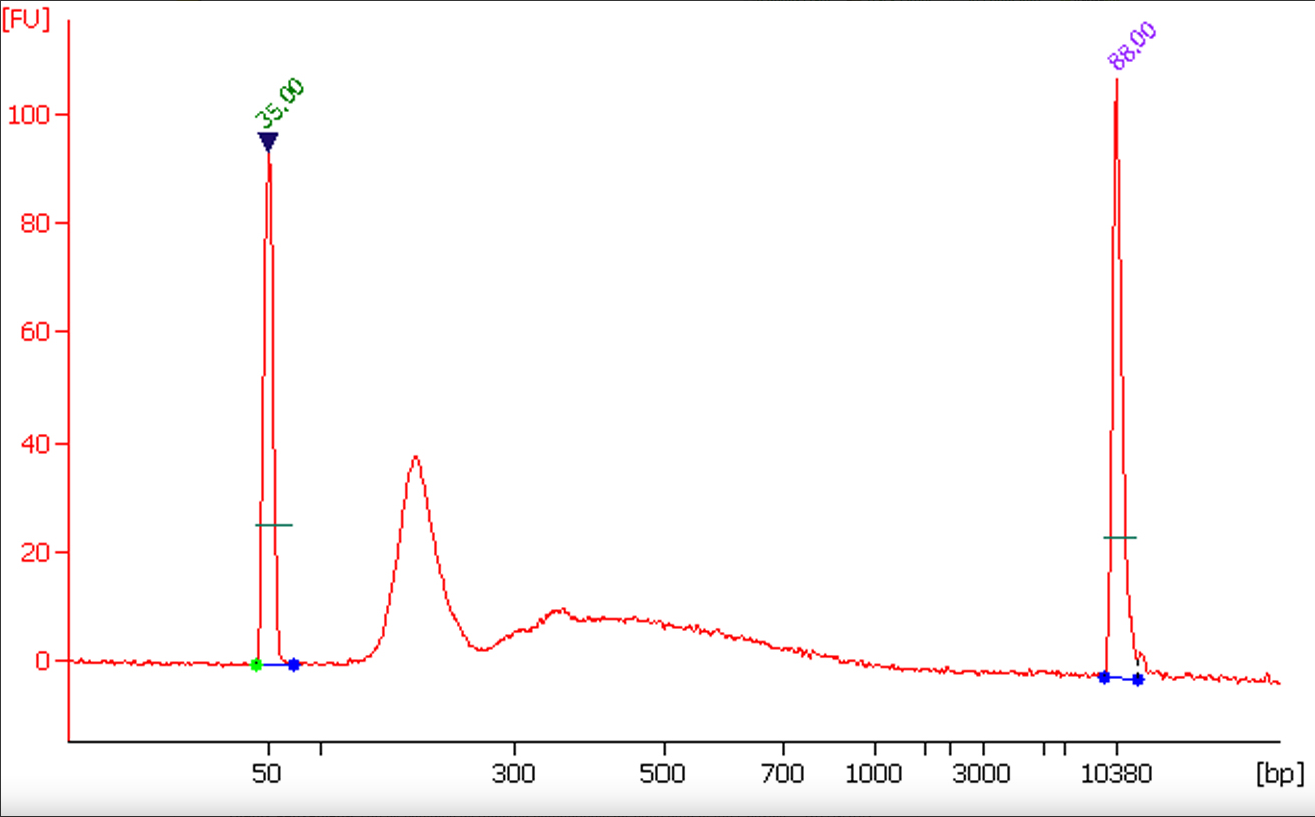
- 超快速、精简程序: 从RNA 到cDNA 文库的过程小于3 小时,动手时间<30 分钟。
- 超级方便: 本试剂盒包含了每一步所需的所有组件,这足以捕获含m6A 的RNA 序列,从而允许以一种很方便的方法来富集,结果可靠一致。
产品组分
|
内容 |
型号:A-P-9018-24(24次) |
型号:A-P-9018-48(48次) |
型号:A-P-9018-96(96次) |
保存条件 |
|
WB (Wash Buffer) |
30ml |
60ml |
120ml |
4°C |
|
ICB (Immuno Capture Buffer) |
5ml |
10ml |
20ml |
常温 |
|
NDE (Nuclear Digestion Enhancer) |
300ul |
600ul |
1200ul |
常温 |
|
CEM (Cleavage Enzyme Mix)* |
60ul |
120ul |
240ul |
–20°C |
|
m6A Antibody (1mg/ml)* |
50 ul |
100ul |
200ul |
–20°C |
|
Non-Immune IgG (1mg/ml) * |
20ul |
40ul |
80ul |
4°C |
|
m6A-Positive Control (200ug/ml) * |
6ul |
12ul |
24ul |
–20°C |
|
PDB (Protein Digestion Buffer) |
5 ml |
10 ml |
20 ml |
常温 |
|
Proteinase K (10mg/ml) * |
100ul |
200ul |
400ul |
4°C |
|
Affinity Beads* |
100ul |
200ul |
400ul |
4°C |
|
RPS (RNA Puritication Solution) |
600ul |
1.2ml |
2.4ml |
4°C |
|
RNA Binding Beads * |
60ul |
120ul |
240ul |
4°C |
|
Elution Buffer |
2000ul |
4000ul |
8000ul |
常温 |
|
使用手册 |
1 |
1 |
1 |
常温 |
文件资源
| 文件下载 | 文件内容 | 资源说明 |
| 产品视频 | A-P-9018-m6A RNA甲基化片段富集试剂盒(qPCR版)【简称:MeRIP试剂盒】 | 操作手册 |
| 有关MeRIP-qPCR引物设计 | 有关MeRIP-qPCR相对表达量计算 | 宣传单页 |
注意事项
| 保存建议【配套现货产品推荐如下】 | 推荐蓝冰或冰袋运输。当您收到产品后,请严格按照说明书建议保存各产品组分。 |
| 如提纯的总RNA是采用trizol溶液的方法 | 推荐:RTSHOW gDNA去除试剂盒【可从提纯的总RNA中有效的去除gDNA,改善qPCR实验数据】 |
| miRNA纯化试剂盒 | 推荐:miRNA纯化试剂盒(R-5007)【可从细胞或组织样本中分离20-200nt的miRNA,siRNA, snRNA等小片段RNA】 |
| mRNA纯化试剂盒 | 推荐:mRNA纯化试剂盒(Z5300)【针对纯化的总RNA来进行】IVDSHOW Oligo(dT)18磁珠(R-9100)【针对细胞和组织样本】 |
| 磁珠法纯化 | 推荐:病毒DNA/RNA提取试剂盒(磁珠法)(SBK186)【针对血清,血浆,鼻拭子,鼻咽拭子,唾液等体液样本来纯化】 |
| RNA纯化试剂盒 | 推荐:总RNA纯化试剂盒(TR01)【据不同的样本来选型,比如纯化病毒RNA选型指南】 |
| 植物样本RNA纯化试剂盒 | 推荐:植物总RNA纯化试剂盒(TR02)【采用离心柱法纯化RNA选型指南】 |
| cDNA反转录试剂盒【pg~ng级别】 | 推荐:IVDSHOW 高保真 RT Plus Master Mix(R-G207)【针对富集的片段化m6A进行反转录成cDNA】 |
| cDNA反转录试剂盒 | 推荐:IVDSHOW 高保真 RT All-in-One Master Mix(R-G208)【针对富集的片段化m6A进行反转录成cDNA】 |
| qPCR试剂盒 | 推荐:EvaGreen qPCR PreMix(R-EMX)【据实时荧光定量PCR仪来选型】 |
| IVDSHOW 16孔合金磁力架 | 推荐:IVDSHOW 16孔合金磁力架(C3111)【可放置16x0.2ml PCR管,非常适合小体积洗脱,少至5ul也可操作】 |
| IVDSHOW 16孔磁力架 | 推荐:IVDSHOW 16孔磁力架(C3001)【可放置16x0.2ml PCR管】快速MS-qPCR试剂盒【甲基化专用酶】 |
| IVDSHOW 96孔磁力架 | 推荐:IVDSHOW 96孔磁力架(C3004)【可放置96孔 PCR板,性价比超高】 |
| m6A兔源多克隆抗体【MeRIP级】 | 推荐:m6A兔源多克隆抗体【MeRIP级】(A-1801)【可做DB,ELISA,IF,IP和MeRIP】 |
| m6A兔源多克隆抗体[2H6]【MeRIP级】 | 推荐:m6A兔源单克隆抗体【MeRIP级】(A-1802)【可做DB,ELISA,IF,IP和MeRIP】 |
FAQ
扩展阅读:CUT&RUN技术是如何帮助提高m6A RNA甲基化免疫沉淀的效果?
Q:N6-methyladenosine (m6A or 6mA)分子式是什么?
A: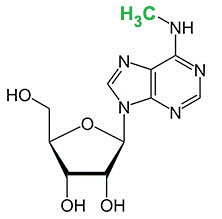 这个就是N6-methyladenosine (m6A)分子式,也就是N6-甲基腺嘌呤,简称:m6A or 6mA。我们也单独出售这种化合物【More】
这个就是N6-methyladenosine (m6A)分子式,也就是N6-甲基腺嘌呤,简称:m6A or 6mA。我们也单独出售这种化合物【More】
Q:CUT&RUN是什么呢?
A:CUT&RUN【Cleavage under targets and release using nuclease】,CUT&RUN具有显著优势,该技术实验周期短,信噪比高,可重复性好,且细胞投入量低,尤其适用于早期胚胎发育、干细胞、肿瘤以及表观遗传学等研究领域。【More】
Q:m6A RNA【RNA中含有的m6A】 是否等同于 6mA or m6A DNA【DNA中含有的6mA or m6A】?
A:m6A RNA和6mA DNA取决于许多因素,包括物种、正常与疾病状态、组织类型(如大脑、肝脏)等。您所掌握的搜索文献可能有助于获得有关其特定样品的更多信息。
Q:如何判定富集下来m6A的效率呢?
A:如果要判断m6A富集效率,我们建议做荧光定量分析,而不是做PCR,因为富集下来的也是RNA。
Q:富集下来的m6A 片段大概在多少呢?引物设计大概在多么大片段合适呢?
A:如果做qPCR,即RT-qPCR,首先将富集下来的m6A片段逆转录为cDNA,富集下来的m6A RNA片段长度约为50-200nts,建议qPCR引物的设计覆盖70-150bps扩增长度,且包含预期有m6A修饰的区域。通常可借助引物设计软件如:Primer3。qPCR的Ct值取决于m6A RNA的富集程度以及逆转录效果,一般来说,用这个试剂盒来富集5ug小鼠脑RNA样本的话Ct值为30-32,阴性对照【Non-immune IgG】的Ct值>35。
Q:能否提供阳性对照的序列?
A:阳性对照的Positive control oligo因为版权原因,是保密的,暂不予提供。
Q:试剂盒提供的阳性对照能做几次?同步实验后的富集百分比大概在多少呢?
A:本试剂盒同步实验够2次使用,每次3ul用量;根据我们的质量控制测试,通常每次阳性对照【200ng/ul,每次3ul用量】的富集效率约为总输入阳性对照【200ng/ul,每次3ul用量】的4-7%。如本试剂盒中每次阳性对照需加3ul,如果按照4-7%计算所得阳性对照富集量大概是:24-42ng/ul这个范围。
Q:关于操作手册中1d-1e中间划线部分的说明?
A:这一步是用来做目的基因IP后qPCR对照的,做qPCR的对照来使用,相当于起内参基因的作用。没有与抗体一起孵育,不用做1a-d,直接酶解。然后进入2d,这个input组加的RPS为25ul。
Q:关于组分m6A-Positive Control 说明?
A:它不包含引物,是一种合成的寡核苷酸,没有种属特异性。主要用于评估试剂盒本身的富集效率的。
Q:在提纯总RNA过程中,是否需要消化DNA?
A:为了尽可能减少DNA中m6A的干扰得到较好的实验结果,我们建议您纯化总RNA时,尽可能采用DNase I【类似的有去除DNA功能的试剂盒如:SpinSHOW 柱上消化试剂盒】对于DNA进行消化处理。或者选购我们针对细胞和组织样本配套的总RNA纯化试剂盒【TR01】其他样本类型可以直接搜索官网查询。
Q:如您采用的是mRNA标本是否可以采用这个盒子呢?
A:本试剂盒的输入样本是提取的RNA,可从任何物种和各种来源(如培养细胞、新鲜/冷冻组织、石蜡包埋组织、血液、体液样本等)中分离出来。纯化的、完整的mRNA也可以作为这个试剂盒的输入材料。RNA应该在无RNase的水或合适的缓冲液(例如,TE)中,并且应该是高质量和相对无DNA的。
Q:如果我采用的是大肠杆菌的样本,是否可以采用这款产品?
A:这款产品我们并非针对此标本专门设计的,暂时还没有验证过。因为这款产品针对的是位点设计的,而非针对物种。一般来说,是可以使用的。
Q:这个富集试剂盒。是只能富集总RNA中的m6A吗?还是说DNA中的m6A也能富集到呢?
A:本试剂盒中提供的m6A抗体可以检测到DNA上的5-mC,因为该抗体对m6A修饰具有特异性。因此,应将纯化的RNA作为起始材料,如本产品指南所述。目前还没有把DNA作为起始材料来富集。
Q:试剂盒中的单独组分:m6A Antibody 是否单独出售?是单克隆抗体,还是多克隆抗体?
A:可以单独定购。型号:A-P-9018-24-m6A 品名:m6A Antibody (1 mg/ml)* 规格:50 µl;试剂盒中的m6A Antibody 是多克隆抗体。也可以选择m6A兔源多克隆抗体【A-1802】和m6A兔源单克隆抗体[2H6]【A-1801】
Q:在MeRIP项目中,为什么Input组和IP组都要做?
MeRIP项目中Input和IP组成一对样品。实验环节IP样品用m6A抗体特异性富集甲基化修饰的RNA片段,而Input仅仅是片段化的RNA则作为对照消减背景噪音,在建库、测序平行开展。结合峰检测分析需整合两个样本的数据,并利用Input数据排除本底表达水平高或非特异性结合的peaks,以提高calling peak的准确性。
【操作层面的注意事项】
Q:试剂盒中的单独组分:ICB是否够用?
A:a).与抗体的反应最多需要189ul ICB(样品+ m6A抗体或非免疫IgG rxns)或191ul ICB(阳性对照寡核苷酸+ m6A抗体rxn)。
b).“Input输入”反应(无抗体对照的rxns)仅需要20ul ICB。
c).因此,对于具有3种条件的8个样品(m6A抗体-189 ul ICB;非免疫IgG-189 ul ICB;“输入”-20 ul ICB),需要24个ICB的总体积 反应(8个样品* 3个条件,每个= 24 rxns)为8 *(189 + 189 + 20)= 3.2 ml,足够了。
d). ICB通常会提供额外的容量(〜0.5 ml)。
Q:针对步骤2b中热循环仪是否盖盖子?
A:对于在没有加热盖子的热循环仪中进行的步骤2b孵化,是指关闭盖子的热量。 如果热循环仪没有这个选项,就在没有盖子的情况下或不盖盖子的情况下,进行孵化反应。
Lin X et. al. (March 2024). Mettl3‑mediated m(6)A RNA methylation regulates osteolysis induced by titanium particles. Mol Med Rep. 29(3)
Liao M et. al. (February 2024). METTL3-mediated m6A modification of NORAD inhibits the ferroptosis of vascular smooth muscle cells to attenuate the aortic dissection progression in an YTHDF2-dependent manner. Mol Cell Biochem.
Zhu M et. al. (February 2024). N(6)-methyladenosine modification of the mRNA for a key gene in purine nucleotide metabolism regulates virus proliferation in an insect vector. Cell Rep. 43(2):113821.
Yang G et. al. (February 2024). Combined GWAS and eGWAS reveals the genetic basis underlying drought tolerance in emmer wheat (Triticum turgidum L.). New Phytol.
Zhao Y et. al. (February 2024). N(6) -methyladenosine mRNA methylation positively regulated the response of poplar to salt stress. Plant Cell Environ.
Wang J et. al. (January 2024). Regulation of ULK1 by WTAP/IGF2BP3 axis enhances mitophagy and progression in epithelial ovarian cancer. Cell Death Dis. 15(1):97.
Tian Y et. al. (January 2024). Identification of m6A-Related Biomarkers in Systemic Lupus Erythematosus: A Bioinformation-Based Analysis. J Inflamm Res. 17:507-526.
Chen H et. al. (January 2024). BMP9-ID1 Pathway Attenuates N(6)-Methyladenosine Levels of CyclinD1 to Promote Cell Proliferation in Hepatocellular Carcinoma. Int J Mol Sci. 25(2)
Zhi D et. al. (December 2023). METTL3/YTHDF1 m(6)A axis promotes tumorigenesis by enhancing DDR2 expression in ovarian cancer. Pathol Res Pract. 253:155047.
Zhang G et. al. (December 2023). Targeting AKT induced Ferroptosis through FTO/YTHDF2-dependent GPX4 m6A methylation up-regulating and degradating in colorectal cancer. Cell Death Discov. 9(1):457.
Zhou Z et. al. (December 2023). METTL3-mediated m(6)A modification of lnc KCNQ1OT1 promotes doxorubicin resistance in breast cancer by regulating miR-103a-3p/MDR1 axis. Epigenetics. 18(1):2217033.
Wang J et. al. (November 2023). HES1 promotes aerobic glycolysis and cancer progression of colorectal cancer via IGF2BP2-mediated GLUT1 m6A modification. Cell Death Discov. 9(1):411.
Hao S et. al. (November 2023). STM2457 Inhibits the Invasion and Metastasis of Pancreatic Cancer by Down-Regulating BRAF-Activated Noncoding RNA N6-Methyladenosine Modification. Curr Issues Mol Biol. 45(11):8852-8863.
Zhao R et. al. (November 2023). The m6A reader IGF2BP2 promotes the progression of esophageal squamous cell carcinoma cells by increasing the stability of OCT4 mRNA. Biochem Cell Biol.
Peng Q et. al. (November 2023). Global m6A methylation and gene expression patterns in human microglial HMC3 cells infected with HIV-1. Heliyon. 9(11):e21307.
Guo Q et. al. (October 2023). The effect on m(6)A methylation writer complex by the reduced MATR3 in pterygium. Acta Histochem. 125(8):152101.
Liu N et. al. (October 2023). The RNA methyltransferase METTL16 enhances cholangiocarcinoma growth through PRDM15-mediated FGFR4 expression. J Exp Clin Cancer Res. 42(1):263.
Li J et. al. (October 2023). Gastric cancer derived exosomal THBS1 enhanced Vγ9Vδ2 T-cell function through activating RIG-I-like receptor signaling pathway in a N6-methyladenosine methylation dependent manner. Cancer Lett. 576:216410.
Shi K et. al. (September 2023). METTL3 exerts synergistic effects on m6A methylation and histone modification to regulate the function of VGF in lung adenocarcinoma. Clin Epigenetics. 15(1):153.
Su X et. al. (August 2023). miR-33a-3p regulates METTL3-mediated AREG stability and alters EMT to inhibit pancreatic cancer invasion and metastasis. Sci Rep. 13(1):13587.
Chen YH et. al. (August 2023). RNA N6-methyladenosine of DHAPAT and PAP involves in regulation of diapause of Bombyx mori via the lipid metabolism pathway. Bull Entomol Res. :1-11.
Wang X et. al. (August 2023). Therapeutic targeting of FUBP3 phase separation by GATA2-AS1 inhibits malate-aspartate shuttle and neuroblastoma progression via modulating SUZ12 activity. Oncogene.
Gu Z et. al. (July 2023). WTAP-mediated m6A modification of IFNE is required for antiviral defense in condyloma acuminata. J Dermatol Sci.
Bei M et. al. (June 2023). Splicing factor TRA2A contributes to esophageal cancer progression via a noncanonical role in lncRNA m(6) A methylation. Cancer Sci.
Liu J et. al. (May 2023). N6-methyladenosine helps Apostichopus japonicus resist Vibrio splendidus infection by targeting coelomocyte autophagy via the AjULK-AjYTHDF/AjEEF-1α axis. Commun Biol. 6(1):547.
Chen J et. al. (May 2023). METTL3 promotes pancreatic cancer proliferation and stemness by increasing stability of ID2 mRNA in a m6A-dependent manner. Cancer Lett. 565:216222.
Xing K et. al. (May 2023). N(6) -methyladenosine mRNA modification regulates transcripts stability associated with cotton fiber elongation. Plant J.
Geng S et. al. (May 2023). METTL3-Mediated m6A Modification of TRIF and MyD88 mRNAs Suppresses Innate Immunity in Teleost Fish, Miichthys miiuy. J Immunol.
Lei C et. al. (May 2023). Hypericin Ameliorates Depression-like Behaviors via Neurotrophin Signaling Pathway Mediating m6A Epitranscriptome Modification. Molecules. 28(9)
Zhong D et. al. (April 2023). Comprehensive analysis of long non-coding RNA expression profiles of GC-1spg cells with m6A methylation knockdown. Gene. :147430.
Li YL et. al. (April 2023). Identification of Metabolism-Related Proteins as Biomarkers of Insulin Resistance and Potential Mechanisms of m(6)A Modification. Nutrients. 15(8)
Cheng S et. al. (March 2023). FTO-mediated m(6)A modification promotes malignant transformation of gastric mucosal epithelial cells in chronic Cag A(+) Helicobacter pylori infection. J Cancer Res Clin Oncol.
Luo S et. al. (March 2023). METTL3-mediated m6A mRNA methylation regulates neutrophil activation through targeting TLR4 signaling. Cell Rep. 42(3):112259.
Chen Y et. al. (February 2023). Methyltransferase-like 3 aggravates endoplasmic reticulum stress in preeclampsia by targeting TMBIM6 in YTHDF2-dependent manner. Mol Med. 29(1):19.
Geng S et. al. (December 2022). FTO promotes innate immunity by controlling NOD1 expression via m(6)A-YTHDF2 manner in teleost. iScience. 25(12):105646.
Ning Z et. al. (December 2022). GMEB2 Promotes the Growth of Colorectal Cancer by Activating ADRM1 Transcription and NF-κB Signalling and Is Positively Regulated by the m(6)A Reader YTHDF1. Cancers (Basel). 14(24)
Bian Y et. al. (December 2022). WTAP dysregulation-mediated HMGN3-m6A modification inhibited trophoblast invasion in early-onset preeclampsia. FASEB J. 36(12):e22617.
Kuang Y et. al. (November 2022). KIAA1429 mediates epithelial mesenchymal transition in sorafenib-resistant hepatocellular carcinoma through m6A methylation modification. Cancer Med.
Jiang T et. al. (November 2022). RNA m6A reader IGF2BP3 promotes metastasis of triple-negative breast cancer via SLIT2 repression. FASEB J. 36(11):e22618.
Hu Z et. al. (September 2022). N6-methyladenosine of Socs1 modulates macrophage inflammatory response in different stiffness environments. Int J Biol Sci. 18(15):5753-5769.
Li W et. al. (August 2022). Comprehensive analysis of RNA m6A methylation in pressure overload-induced cardiac hypertrophy. BMC Genomics. 23(1):576.
Chen D. et. al. (June 2022). N6-methyladenosine methylation analysis reveals transcriptome-wide expression response to salt stress in rice roots Environmental and Experimental Botany. 201
Wang X et. al. (May 2022). SRSF9 promotes colorectal cancer progression via stabilizing DSN1 mRNA in an m6A-related manner. J Transl Med. 20(1):198.
Xu C et. al. (April 2022). Methyltransferase-Like 3 Rescues the Amyloid-beta protein-Induced Reduction of Activity-Regulated Cytoskeleton Associated Protein Expression <i>via</i> YTHDF1-Dependent N6-Methyladenosine Modification. Front Aging Neurosci. 14:890134.
Ji et. al. (April 2022). ALKBH5-induced circRNA NRIP1 promotes glycolysis in thyroid cancer cells by targeting the miR-541-5p/PKM2 and miR-3064-5p/PKM2 axes Research Square.
Chen et. al. (April 2022). Transcriptome-wide N6-methyladenosine methylome profiling of Bombyx mori reveals a potential mechanism of epigenetic regulation on diapause Research Square.
Zhou T et. al. (March 2022). Direct Full-Length RNA Sequencing Reveals an Important Role of Epigenetics During Sexual Reversal in Chinese Soft-Shelled Turtle. Front Cell Dev Biol. 10:876045.
Huang J et. al. (January 2022). FTO suppresses glycolysis and growth of papillary thyroid cancer via decreasing stability of APOE mRNA in an N6-methyladenosine-dependent manner. J Exp Clin Cancer Res. 41(1):42.
Xue-yin Pan et. al. (December 2021). METTL3 facilitates hepatic fibrosis progression via m6A-YTHDF2 dependent silencing of GPR161 bioRxiv.
Zhao et. al. Hyperactivity of the CD155 immune checkpoint suppresses anti-vir Nat Cardiovasc Res. 1:634–648.
免责申明:本网站销售的所有产品均不得用于人类或动物之临床诊断或治疗,仅可用于工业或者科研等非医疗目的。(获得国家相关部门批准的产品除外)
版权所有:艾维缔科技怀来有限公司 备案号:冀ICP备20011415号